差异脂质分类富集分析
脂质代谢途径研究计划(Lipid metabolites and pathways strategy, LIPID MAPS)是一个关于脂质分类、功能注释、代谢途径的数据库。[1],各个数据库中包含了大量的有用信息:基因组信息存储在GENES数据库里,包括完整和部分测序的基因组序列;更高级的功能信息存储在PATHWAY数据库里,包括图解的细胞生化过程如代谢、膜转运、信号传递、细胞周期,还包括同系保守的子通路等信息;KEGG的另一个数据 库是LIGAND,包含关于化学物质、酶分子、酶反应等信息。
通过LipidMaps对差异脂质进行结构、类别等信息注释,并进行分类富集分析,根据分类富集分析的结果可进一步了解脂质的功能与代谢途径。
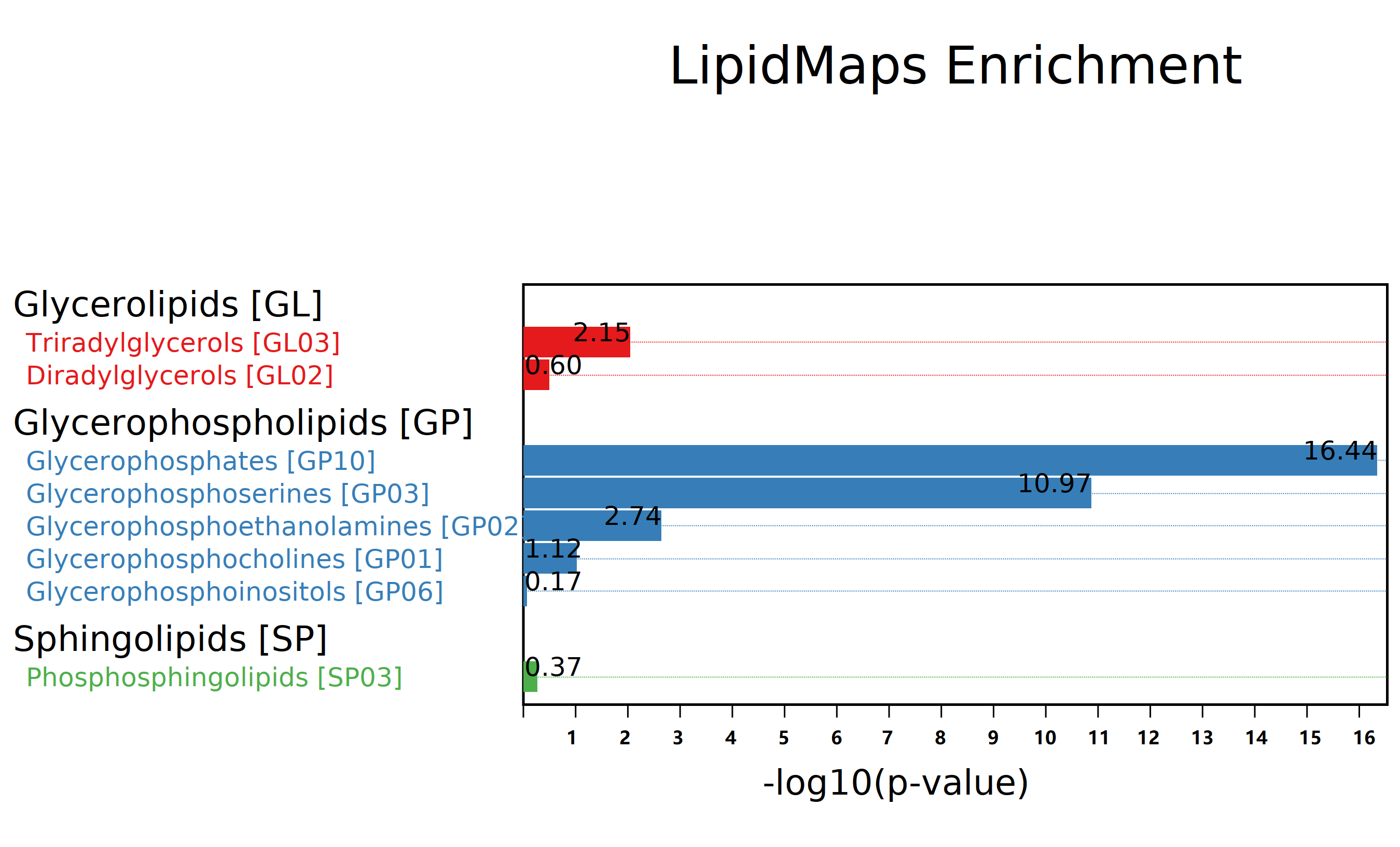
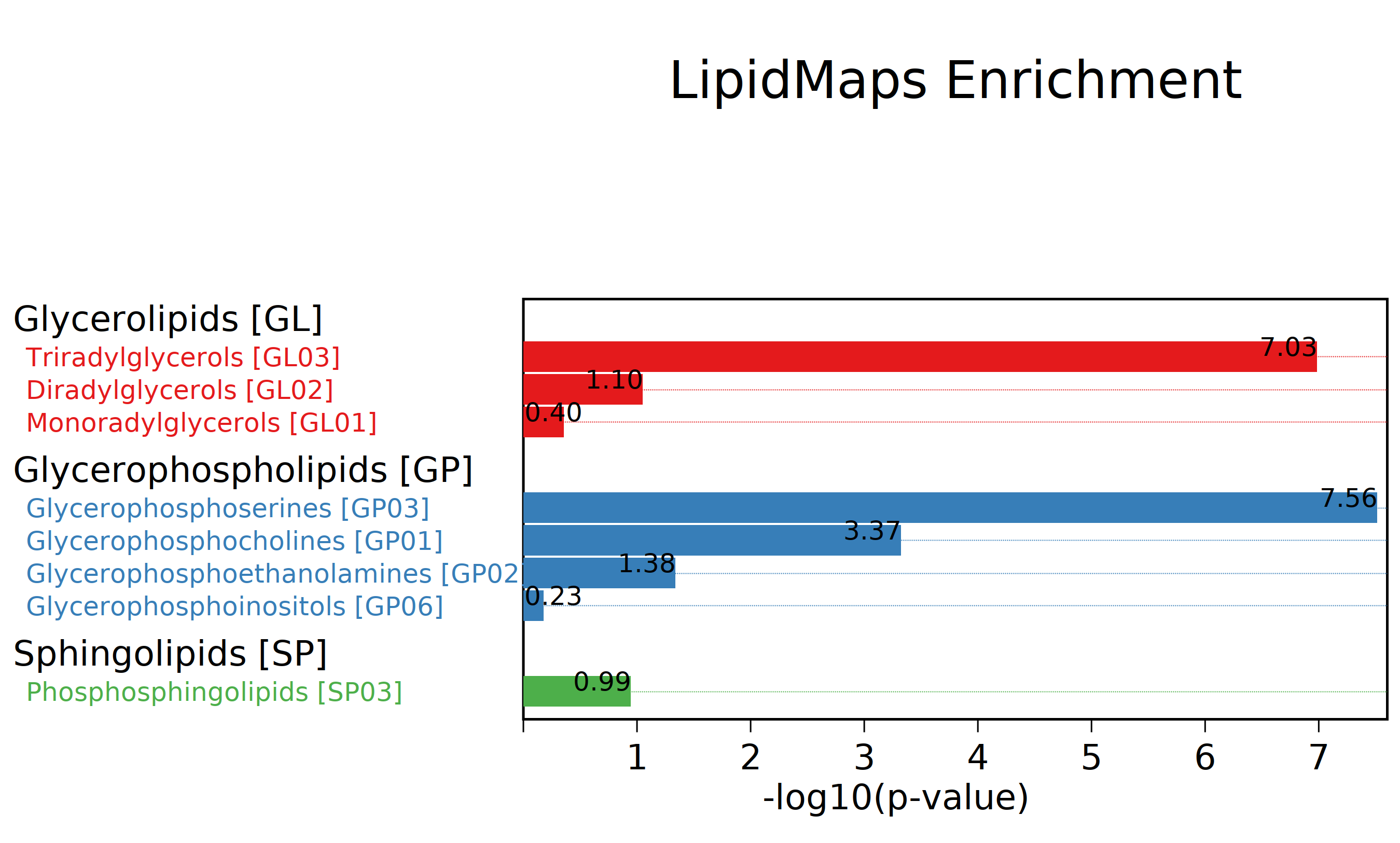
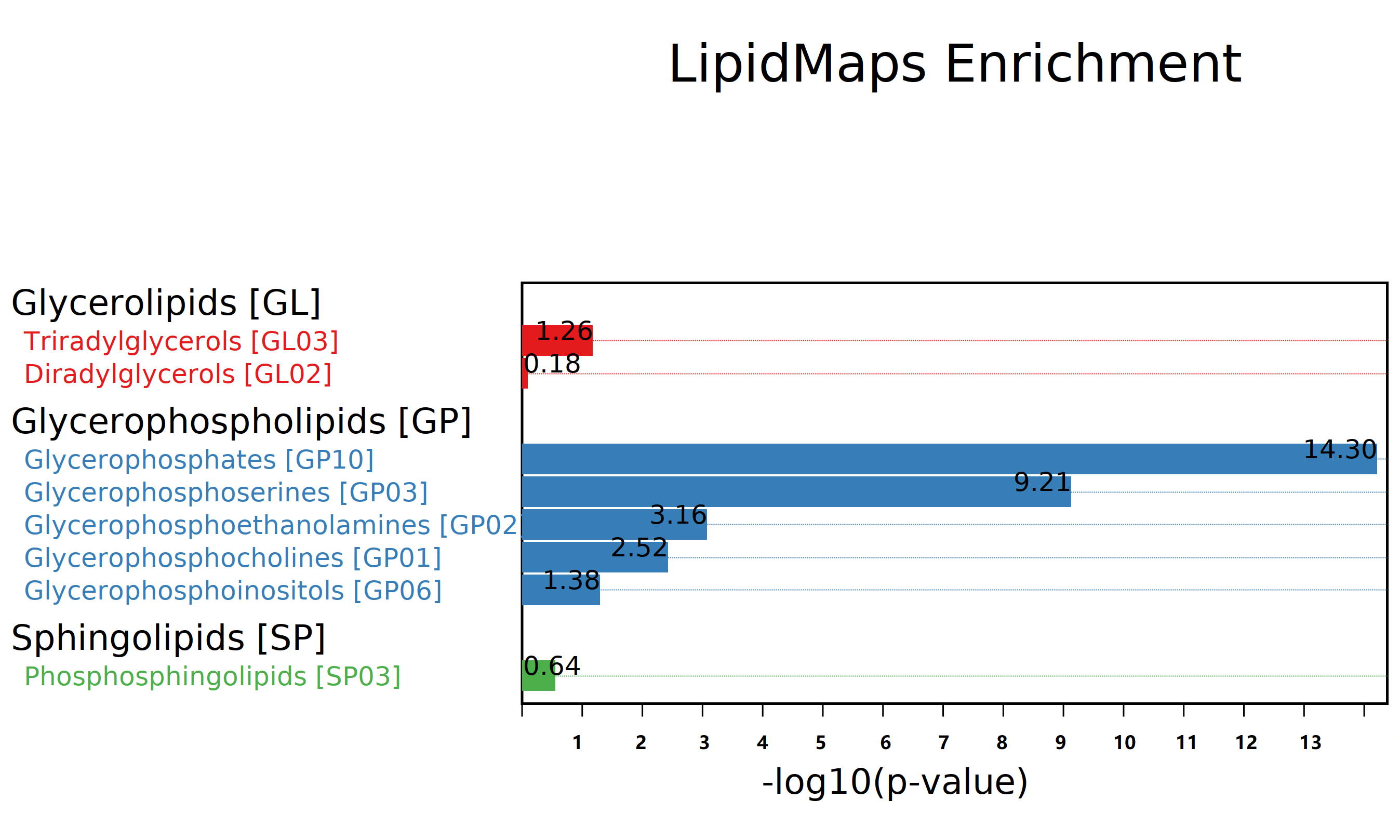
差异脂质功能分类富集图:基于lipidmaps根据脂质的功能相似性,对脂质进行分类,并对各个类别进行富集分析,对不同脂质在同一功能分类中的数量进行fisher检验得到对应富集显著程度的 p value值 ,-log10 p value数值越大,表示在该功能分类中富集到的脂质越多。图中不同颜色表示不用的功能分类,-log10 p value数值越大表示富集显著性越高。
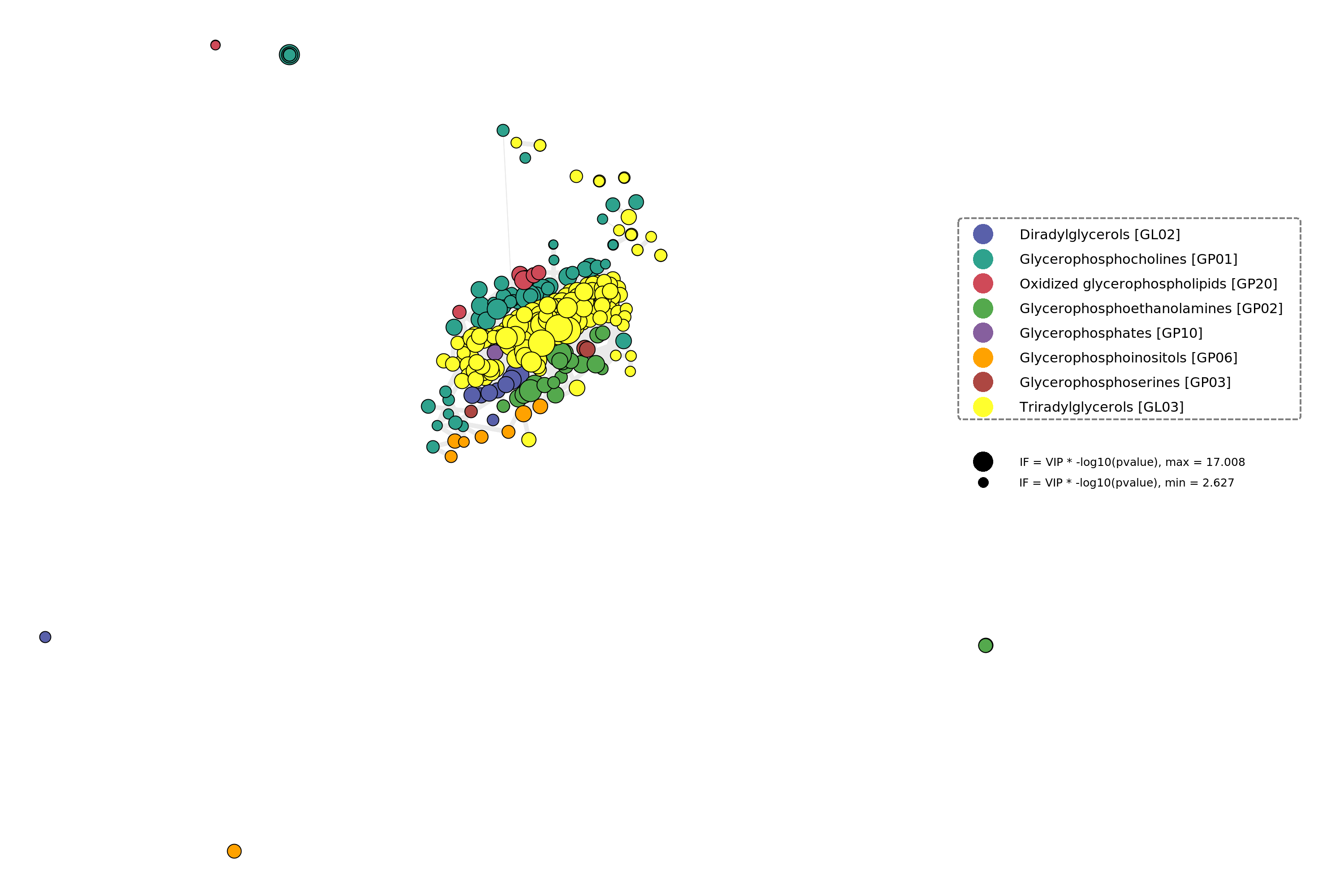
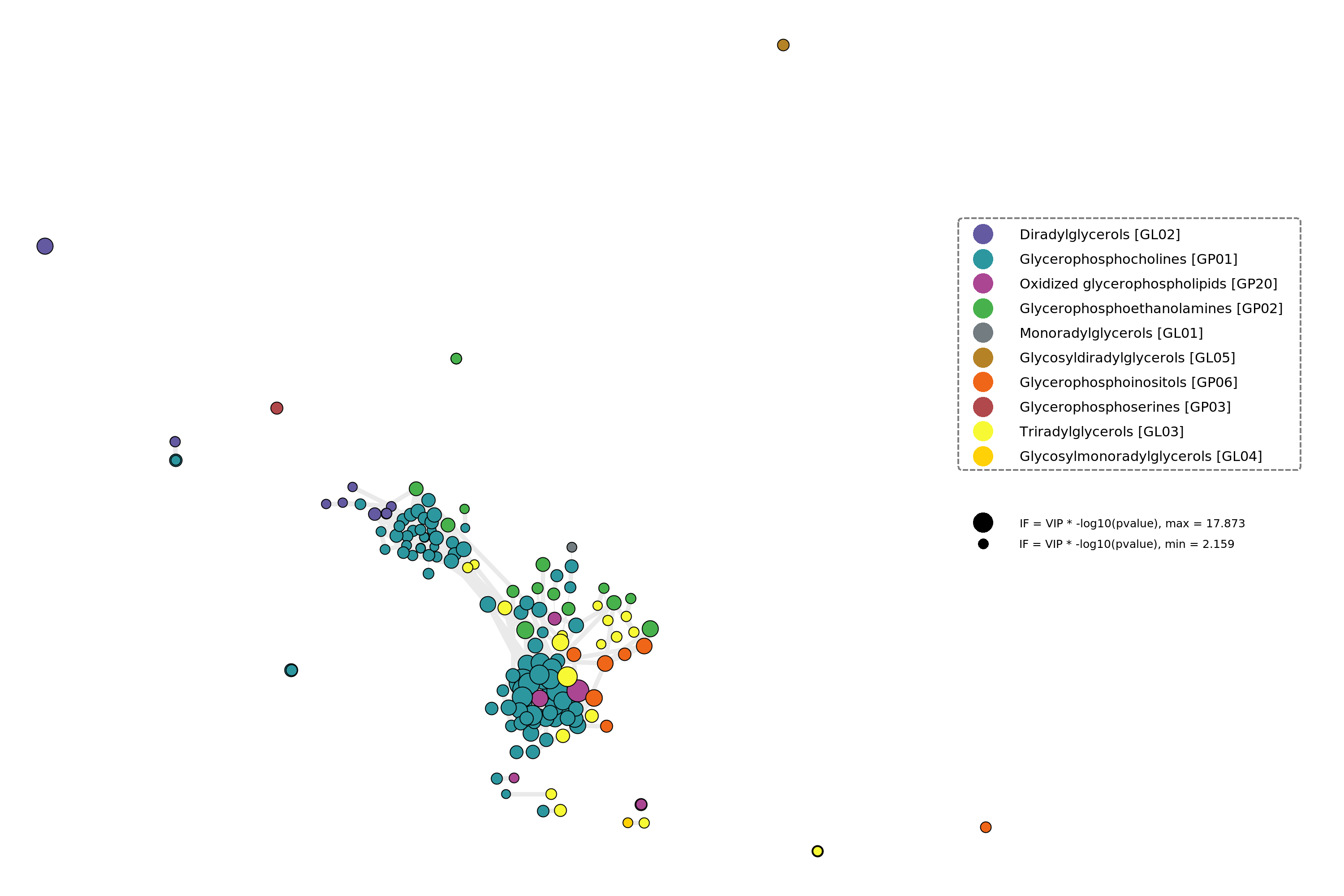
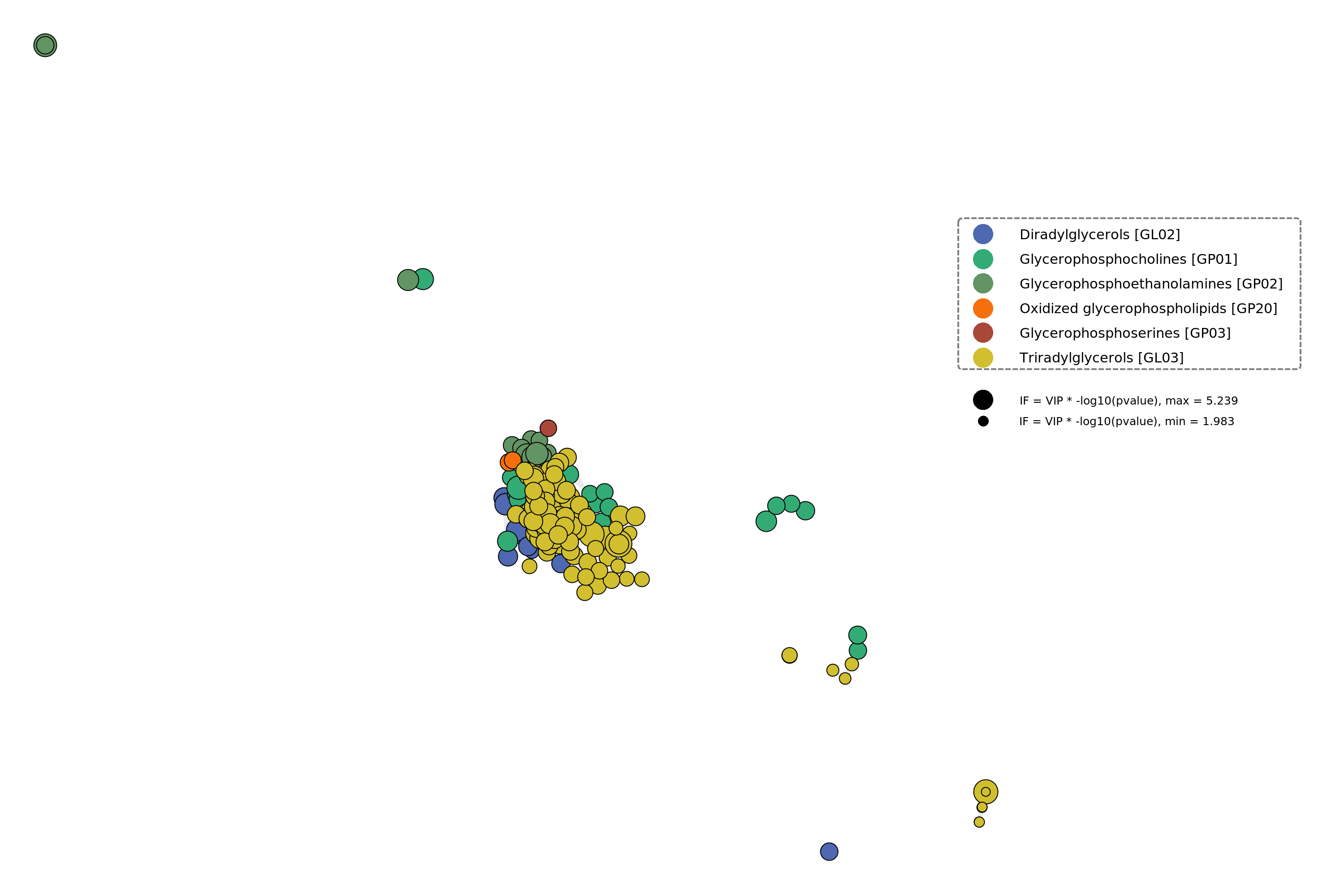
差异脂质关联网络图:计算不同脂质间相对峰响应值的皮尔森相关系数构建关联网络,不同的颜色代表脂质不同的功能分类,圆圈大小代表IF数值,数值越大,脂质的差异越大。圆圈之间的线条粗细代表相关性的程度,皮尔森相关度的绝对值越高,线条越粗,一个点表示一个脂质。
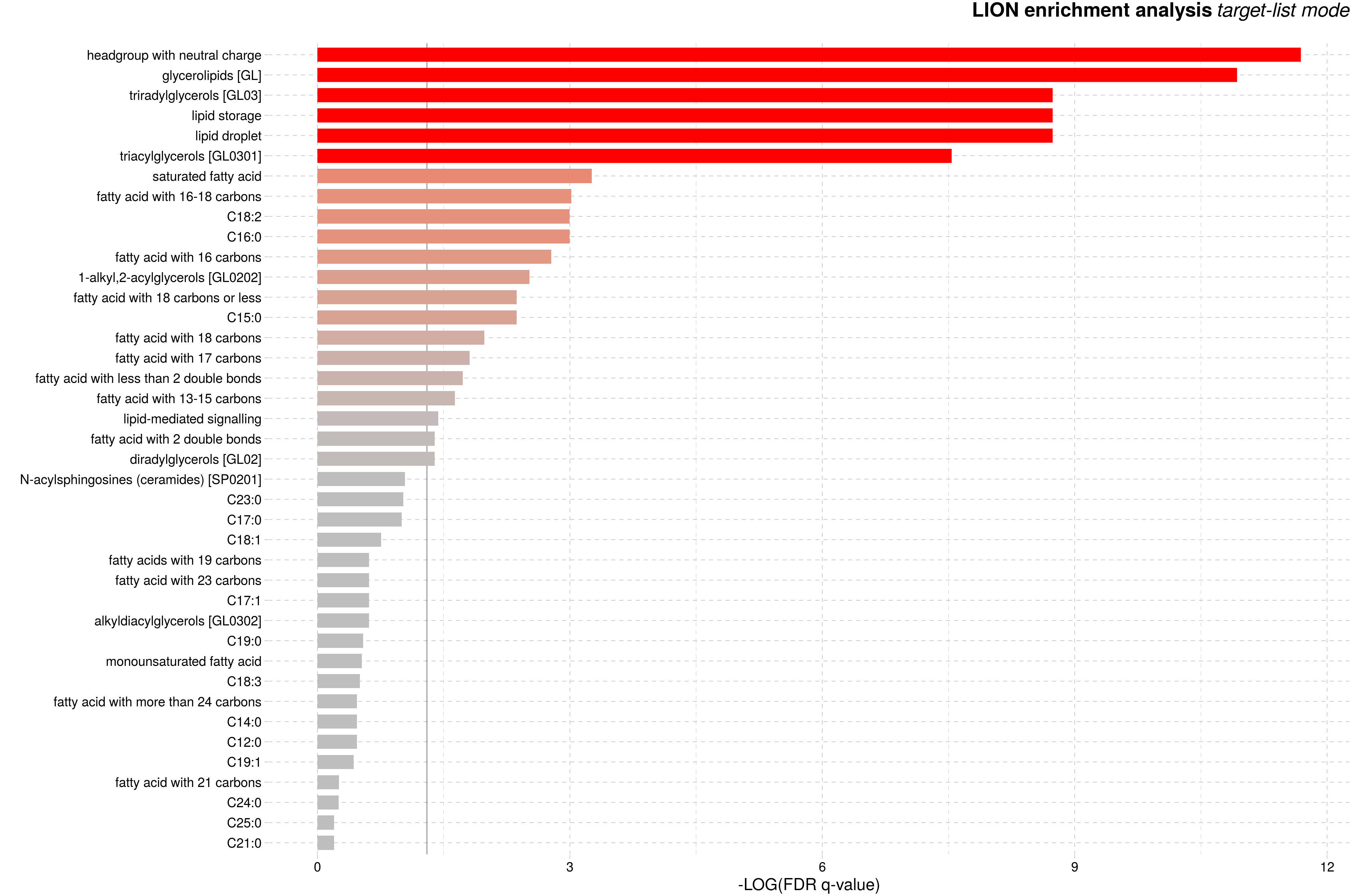
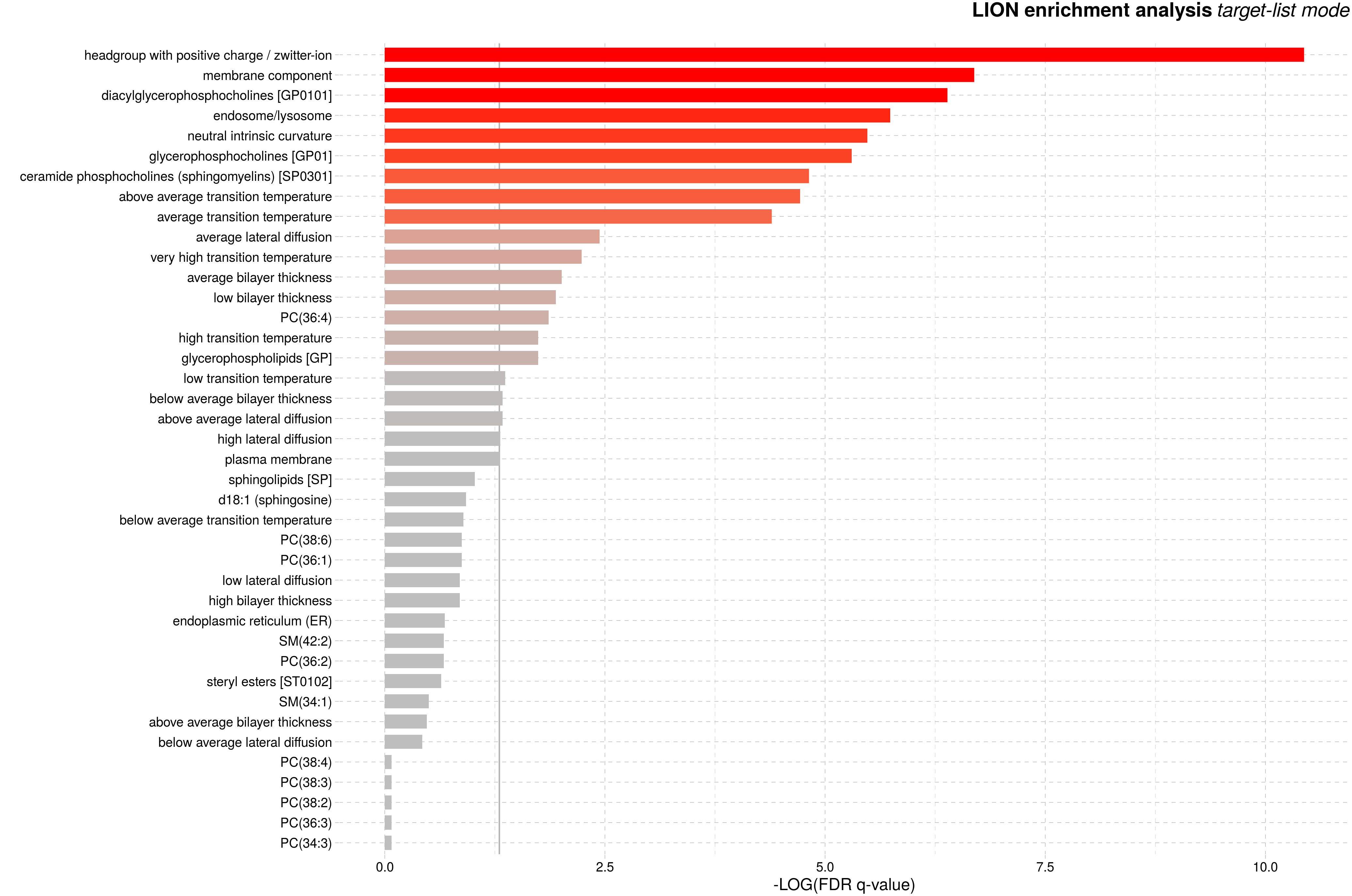
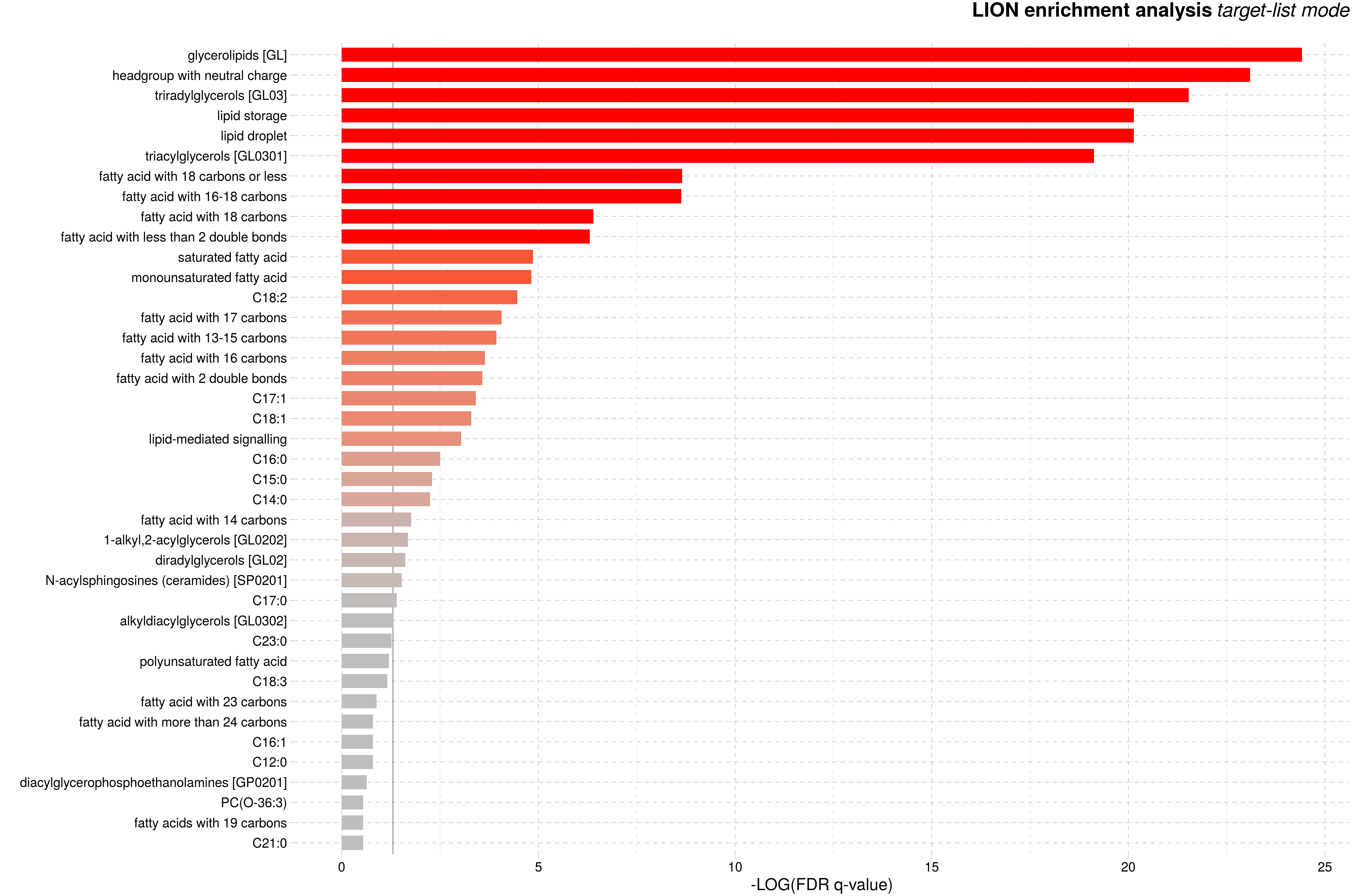
差异脂质结构注释富集图:根据脂质的结构相似性对已有注释信息的脂质结构进行分类,并对各个类别进行富集分析。横坐标代表错误发现率(false discovery rate, FDR)校正后的q value,再经log10取对数后的负值,即-log10 (FDR q value),其中点线代表显著性富集的阈值(q <0.05)。条形图越长,颜色越红表示富集结果越显著。
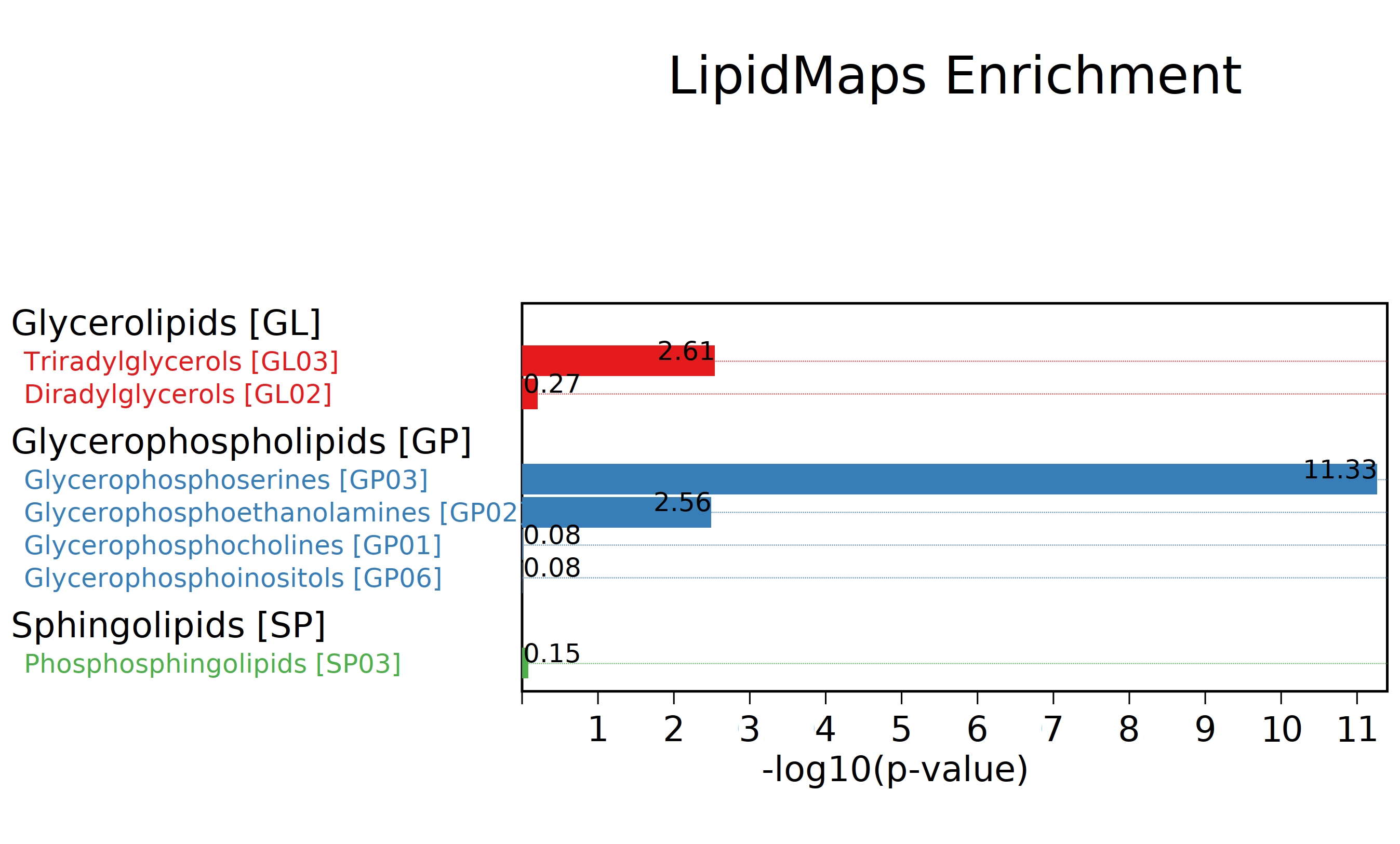
图:差异脂质功能分类富集图

图:差异脂质关联网络图
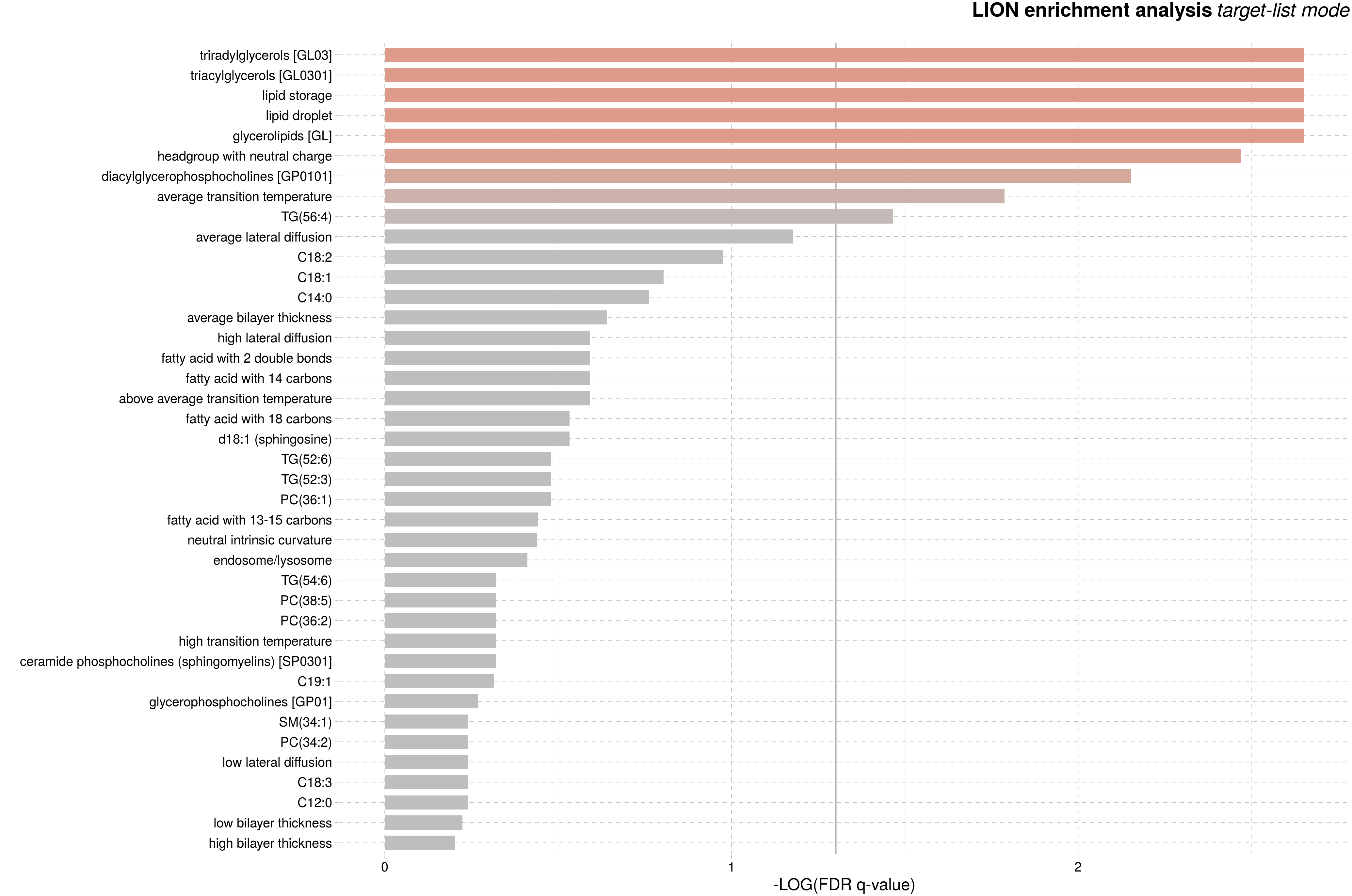
图:差异脂质结构注释富集图
| term | name | description | score | pvalue | FDR | cluster | enriched |
|---|---|---|---|---|---|---|---|
| GP03 | Glycerophosphoserines [GP03] | Glycerophosphoserines [GP03] | 0.0113122171945701 | 4.64563215149972E-12 | 1.53305860999491E-10 | 442 | 5/394 |
| GP02 | Glycerophosphoethanolamines [GP02] | Glycerophosphoethanolamines [GP02] | 0.056140350877193 | 0.00273767361572277 | 0.0301144097729505 | 570 | 32/367 |
| GL03 | Triradylglycerols [GL03] | Triradylglycerols [GL03] | 0.0594059405940594 | 0.00247625754113989 | 0.0408582494288082 | 707 | 42/357 |
| FA01 | Fatty Acids and Conjugates [FA01] | Fatty Acids and Conjugates [FA01] | 0 | 1 | 1.60975609756098 | 32 | 0/399 |
| FA02 | Octadecanoids [FA02] | Octadecanoids [FA02] | 0 | 1 | 1.60975609756098 | 11 | 0/399 |
| FA03 | Eicosanoids [FA03] | Eicosanoids [FA03] | 0 | 1 | 1.60975609756098 | 41 | 0/399 |
| FA07 | Fatty esters [FA07] | Fatty esters [FA07] | 0 | 1 | 1.60975609756098 | 243 | 0/399 |
| FA04 | Docosanoids [FA04] | Docosanoids [FA04] | 0 | 1 | 1.60975609756098 | 2 | 0/399 |
| FA05 | Fatty alcohols [FA05] | Fatty alcohols [FA05] | 0 | 1 | 1.60975609756098 | 57 | 0/399 |
| FA06 | Fatty aldehydes [FA06] | Fatty aldehydes [FA06] | 0 | 1 | 1.60975609756098 | 22 | 0/399 |
| FA08 | Fatty amides [FA08] | Fatty amides [FA08] | 0 | 1 | 1.60975609756098 | 26 | 0/399 |
| GL01 | Monoradylglycerols [GL01] | Monoradylglycerols [GL01] | 0 | 1 | 1.60975609756098 | 9 | 0/399 |
| GP00 | Other Glycerophospholipids [GP00] | Other Glycerophospholipids [GP00] | 0 | 1 | 1.60975609756098 | 1 | 0/399 |
| GP04 | Glycerophosphoglycerols [GP04] | Glycerophosphoglycerols [GP04] | 0 | 1 | 1.60975609756098 | 101 | 0/399 |
| GP05 | Glycerophosphoglycerophosphates [GP05] | Glycerophosphoglycerophosphates [GP05] | 0 | 1 | 1.60975609756098 | 2 | 0/399 |
| GP07 | Glycerophosphoinositol monophosphates [GP07] | Glycerophosphoinositol monophosphates [GP07] | 0 | 1 | 1.60975609756098 | 1 | 0/399 |
| GP10 | Glycerophosphates [GP10] | Glycerophosphates [GP10] | 0 | 1 | 1.60975609756098 | 465 | 0/399 |
| GP12 | Glycerophosphoglycerophosphoglycerols [GP12] | Glycerophosphoglycerophosphoglycerols [GP12] | 0 | 1 | 1.60975609756098 | 394 | 0/399 |
| GP13 | CDP-Glycerols [GP13] | CDP-Glycerols [GP13] | 0 | 1 | 1.60975609756098 | 35 | 0/399 |
| SP01 | Sphingoid bases [SP01] | Sphingoid bases [SP01] | 0 | 1 | 1.60975609756098 | 5 | 0/399 |
| SP06 | Acidic glycosphingolipids [SP06] | Acidic glycosphingolipids [SP06] | 0 | 1 | 1.60975609756098 | 1 | 0/399 |
| ST05 | Steroid conjugates [ST05] | Steroid conjugates [ST05] | 0 | 1 | 1.60975609756098 | 12 | 0/399 |
| ST04 | Bile acids and derivatives [ST04] | Bile acids and derivatives [ST04] | 0 | 1 | 1.60975609756098 | 2 | 0/399 |
| ST02 | Steroids [ST02] | Steroids [ST02] | 0 | 1 | 1.60975609756098 | 14 | 0/399 |
| ST01 | Sterols [ST01] | Sterols [ST01] | 0 | 1 | 1.60975609756098 | 25 | 0/399 |
| PR01 | Isoprenoids [PR01] | Isoprenoids [PR01] | 0 | 1 | 1.60975609756098 | 2 | 0/399 |
| PR02 | Quinones and hydroquinones [PR02] | Quinones and hydroquinones [PR02] | 0 | 1 | 1.60975609756098 | 1 | 0/399 |
| SL01 | Acylaminosugars [SL01] | Acylaminosugars [SL01] | 0 | 1 | 1.60975609756098 | 1 | 0/399 |
| PK12 | Flavonoids [PK12] | Flavonoids [PK12] | 0 | 1 | 1.60975609756098 | 1 | 0/399 |
| GP06 | Glycerophosphoinositols [GP06] | Glycerophosphoinositols [GP06] | 0.0945945945945946 | 0.840437358064957 | 3.96206183087765 | 74 | 7/392 |
| GL02 | Diradylglycerols [GL02] | Diradylglycerols [GL02] | 0.0784313725490196 | 0.536250665086374 | 4.42406798696258 | 306 | 24/375 |
| GP01 | Glycerophosphocholines [GP01] | Glycerophosphocholines [GP01] | 0.0881953867028494 | 0.833926083957073 | 4.5865934617639 | 737 | 65/334 |
| SP03 | Phosphosphingolipids [SP03] | Phosphosphingolipids [SP03] | 0.1 | 0.706507643043685 | 4.66295044408832 | 20 | 2/397 |
注:term,脂质类别编号;name,脂质分类名称;description,脂质分类描述;score:富集分析值;pvalue,超几何分布检验的p值;FDR,假阳性校正后值;cluster,聚类结果数值;enriched,富集脂质数量/该类总数量;IDs,富集到的脂质LipidMapID。