差异代谢物通路分析
KEGG(Kyoto Encyclopedia of Genes and Genomes)是一个整合了基因组、化学和系统功能信息的数据库[1],各个数据库中包含了大量的有用信息:基因组信息存储在GENES数据库里,包括完整和部分测序的基因组序列;更高级的功能信息存储在PATHWAY数据库里,包括图解的细胞生化过程如代谢、膜转运、信号传递、细胞周期,还包括同系保守的子通路等信息;KEGG的另一个数据 库是LIGAND,包含关于化学物质、酶分子、酶反应等信息。
KEGG提供的整合代谢途径(pathway)查询十分出色,包括碳水化合物、核苷、氨基酸等的代谢及有机物的生物降解,不仅提供了所有可能的代谢途径,而且对催化各步反应的酶进行了全面的注解,包括氨基酸序列、PDB库的链接等。KEGG是进行生物体内代谢分析、代谢网络研究的强有力工具,它有助于研究者把基因及其表达信息作为一个整体网络进行研究。
MetPA是metaboanalyst(www .metaboanalyst.ca)的一部分,主要基于KEGG代谢通路。MetPA数据库通过代谢通路浓缩和拓扑分析,识别出可能的受生物扰动的代谢通路,进而对代谢物的代谢通路进行分析。采用MetPA数据库可以分析两组差异代谢物的相关代谢通路,采用的数据分析算法是超几何检验,pathway拓扑结构采用的是Relative-betweeness Centrality。
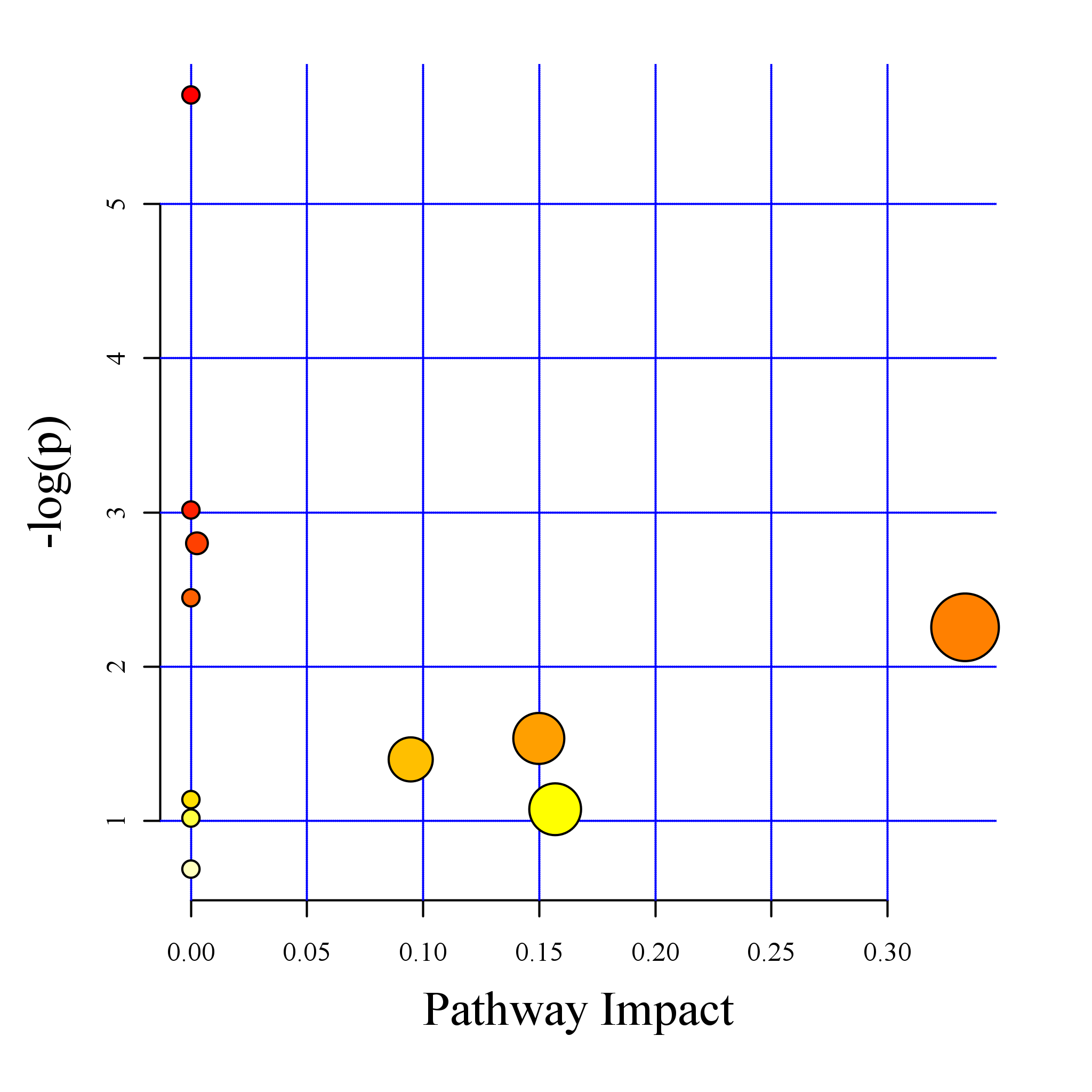
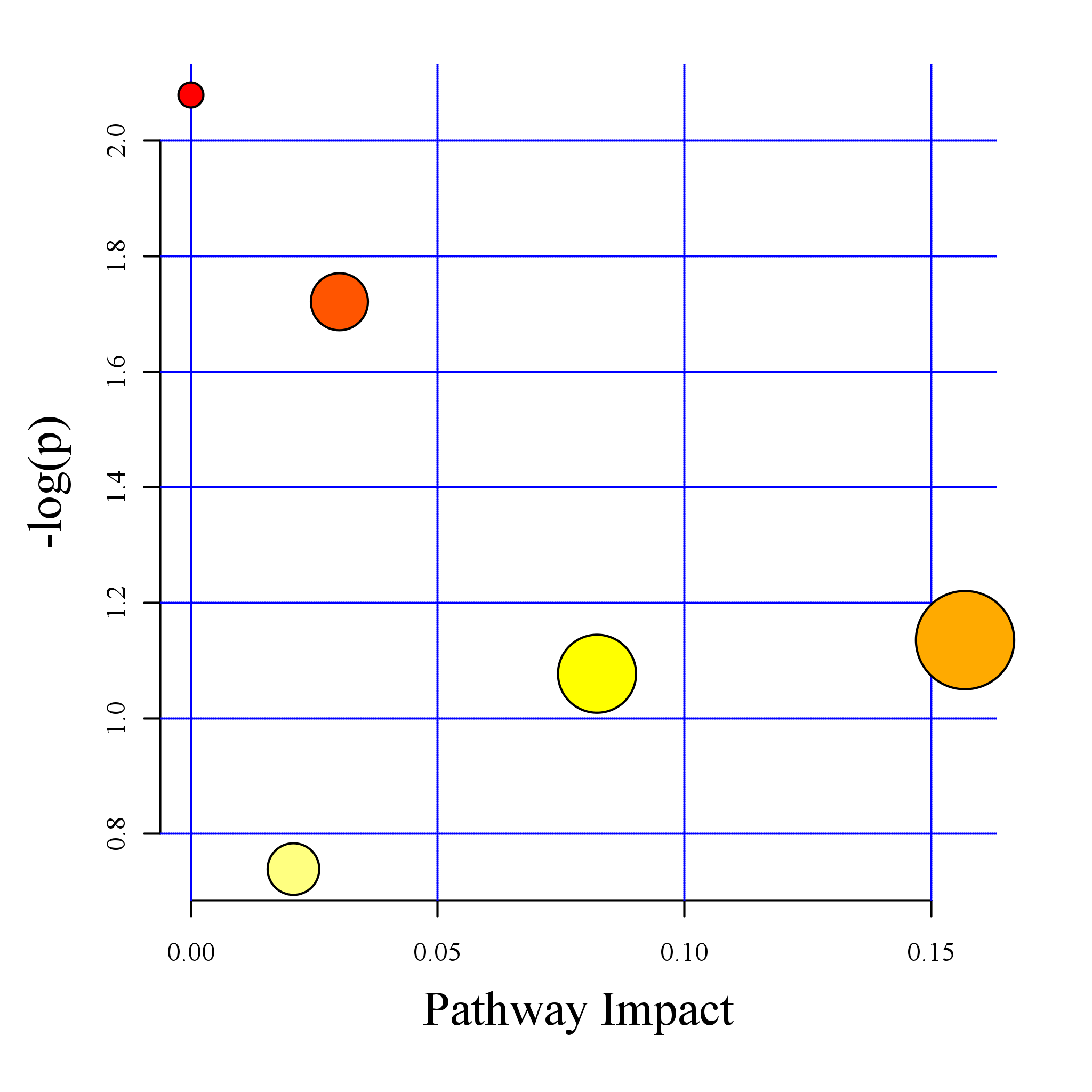
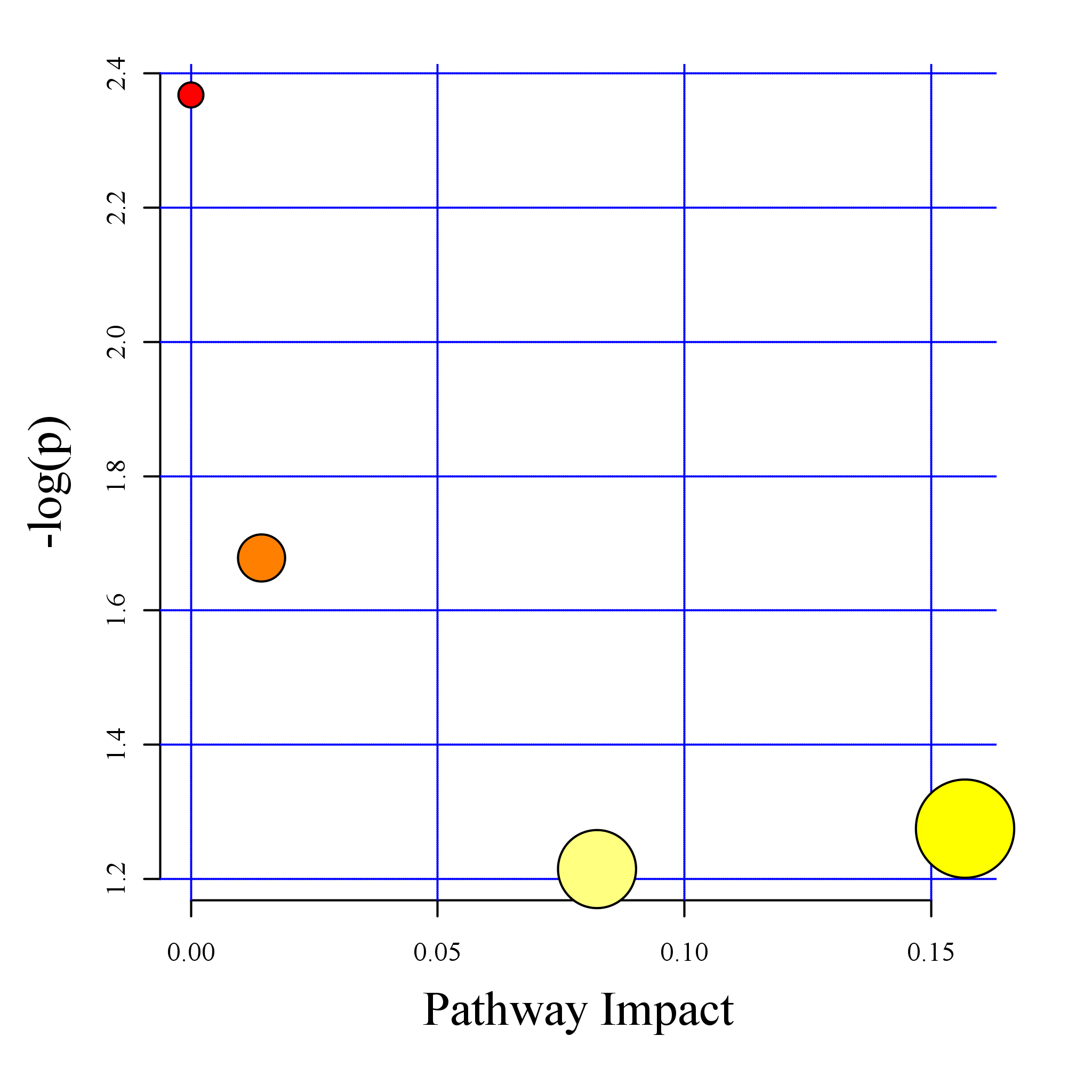
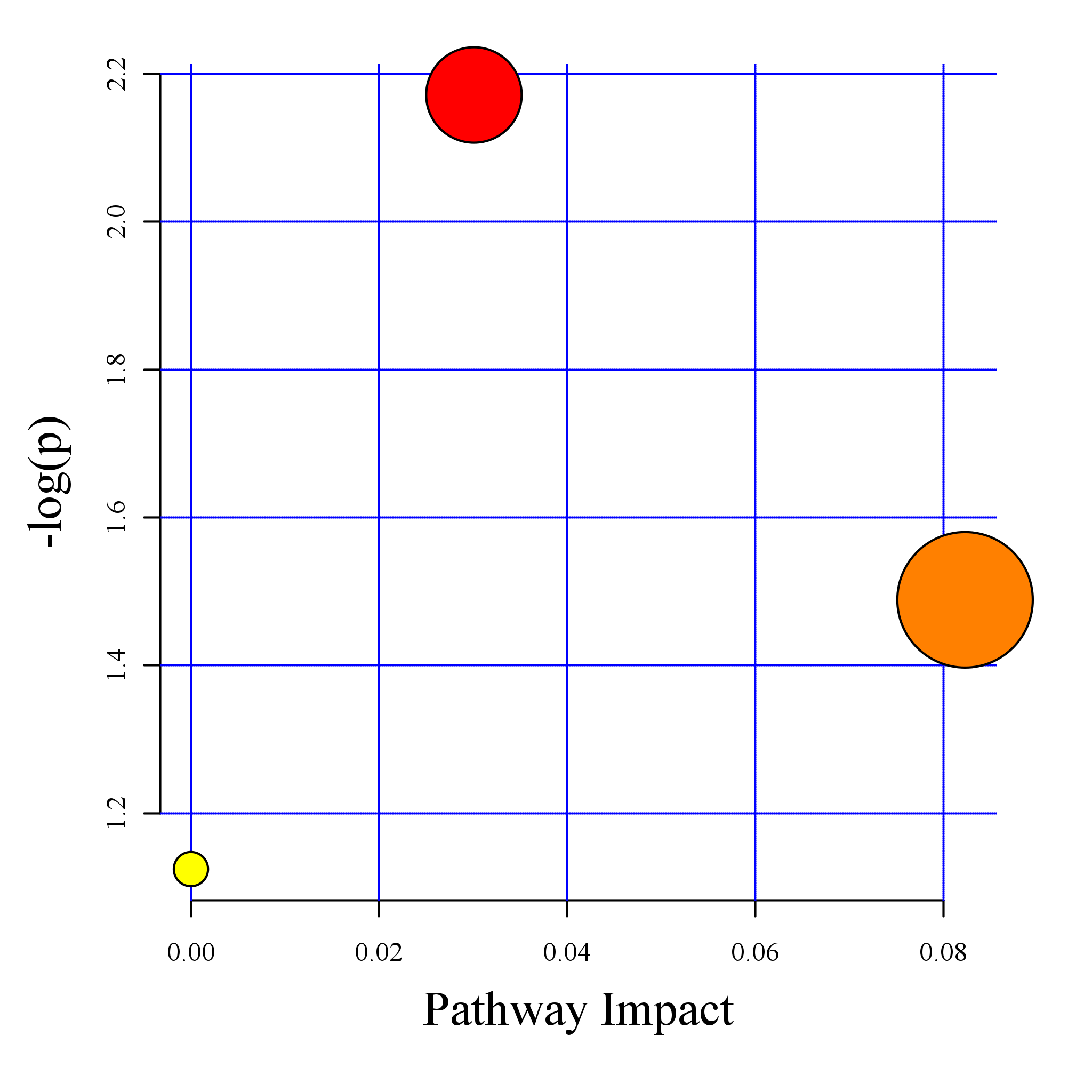
图:代谢通路影响因子图
| Total | Hits | Raw p | -log(p) | Holm adjust | FDR | Impact | compounds | pathway | |
|---|---|---|---|---|---|---|---|---|---|
| Aminoacyl-tRNA biosynthesis | 67 | 4 | 0.0033212 | 5.7074 | 0.26902 | 0.26902 | 0 | C00064;C00073;C00123;C00078 | rno00970 |
| D-Glutamine and D-glutamate metabolism | 5 | 1 | 0.04901 | 3.0157 | 1 | 1 | 0 | C00064 | rno00471 |
| Pyrimidine metabolism | 41 | 2 | 0.060817 | 2.7999 | 1 | 1 | 0.0026 | C00064;C00881 | rno00240 |
| Nitrogen metabolism | 9 | 1 | 0.086602 | 2.4464 | 1 | 1 | 0 | C00064 | rno00910 |
| Valine; leucine and isoleucine biosynthesis | 11 | 1 | 0.10488 | 2.255 | 1 | 1 | 0.33333 | C00123 | rno00290 |
| Alanine; aspartate and glutamate metabolism | 24 | 1 | 0.21563 | 1.5342 | 1 | 1 | 0.14979 | C00064 | rno00250 |
| Cysteine and methionine metabolism | 28 | 1 | 0.24705 | 1.3981 | 1 | 1 | 0.09464 | C00073 | rno00270 |
| Valine; leucine and isoleucine degradation | 38 | 1 | 0.32058 | 1.1376 | 1 | 1 | 0 | C00123 | rno00280 |
| Tryptophan metabolism | 41 | 1 | 0.3413 | 1.075 | 1 | 1 | 0.15684 | C00078 | rno00380 |
| Arginine and proline metabolism | 44 | 1 | 0.36143 | 1.0177 | 1 | 1 | 0 | C00064 | rno00330 |
| Purine metabolism | 68 | 1 | 0.50311 | 0.68695 | 1 | 1 | 0 | C00064 | rno00230 |
注:Total,目标代谢通路中代谢物的总数;Hits,目标代谢通路中差异代谢物数量,Raw p,超几何分布检验的p值;-log(p):对p值的自然对数取负值;Holm adjust,Holm假阳性矫正后p值;FDR,假阳性校正后值;Impact,代谢通路影响值;compounds,代谢物KEGG ID;pathway,代谢物代谢通路ID