差异代谢物通路分析
KEGG(Kyoto Encyclopedia of Genes and Genomes)是一个整合了基因组、化学和系统功能信息的数据库[1],各个数据库中包含了大量的有用信息:基因组信息存储在GENES数据库里,包括完整和部分测序的基因组序列;更高级的功能信息存储在PATHWAY数据库里,包括图解的细胞生化过程如代谢、膜转运、信号传递、细胞周期,还包括同系保守的子通路等信息;KEGG的另一个数据 库是LIGAND,包含关于化学物质、酶分子、酶反应等信息。
KEGG提供的整合代谢途径(pathway)查询十分出色,包括碳水化合物、核苷、氨基酸等的代谢及有机物的生物降解,不仅提供了所有可能的代谢途径,而且对催化各步反应的酶进行了全面的注解,包括氨基酸序列、PDB库的链接等。KEGG是进行生物体内代谢分析、代谢网络研究的强有力工具,它有助于研究者把基因及其表达信息作为一个整体网络进行研究。
MetPA是metaboanalyst(www .metaboanalyst.ca)的一部分,主要基于KEGG代谢通路。MetPA数据库通过代谢通路浓缩和拓扑分析,识别出可能的受生物扰动的代谢通路,进而对代谢物的代谢通路进行分析。采用MetPA数据库可以分析两组差异代谢物的相关代谢通路,采用的数据分析算法是超几何检验,pathway拓扑结构采用的是Relative-betweeness Centrality。
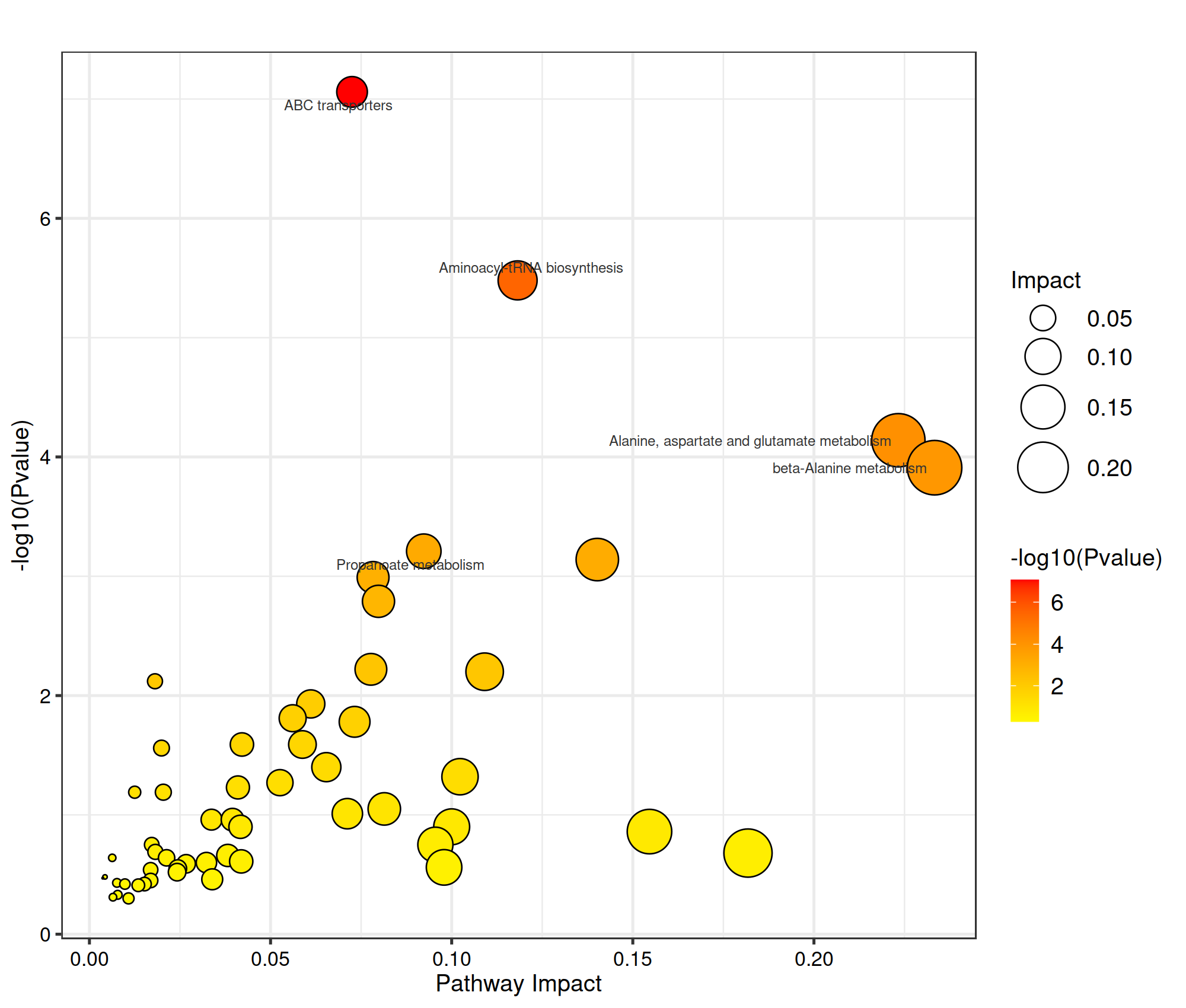
图:代谢通路影响因子图
DOWNLOAD
| pathway_id | pathway_name | Total | Hits | Up_hits | Down_hits | Pvalue | -Log10(Pvalue) | FDR | Impact | compounds | Compound_name |
|---|---|---|---|---|---|---|---|---|---|---|---|
| osa02010 | ABC transporters | 138 | 10 | 9 | 1 | 8.74E-08 | 7.058281873 | 9.27E-06 | 0.0725 | C00037; C00049; C00079; C00116; C00123; C00134; C00137; C00188; C00299; C00387 | Glycine; L-Aspartic acid; DL-Phenylalanine; Glycerol; L-Leucine; Putrescine; Myo-Inositol; L-Threonine; Uridine; Guanosine |
| osa00970 | Aminoacyl-tRNA biosynthesis | 52 | 6 | 6 | 0 | 3.28E-06 | 5.484782872 | 0.000173577 | 0.1182 | C00037; C00049; C00073; C00079; C00123; C00188 | Glycine; L-Aspartic acid; L-Methionine; DL-Phenylalanine; L-Leucine; L-Threonine |
| osa00410 | beta-Alanine metabolism | 32 | 4 | 2 | 2 | 0.000124326 | 3.905437335 | 0.003294644 | 0.2333 | C00049; C00099; C00334; C01013 | L-Aspartic acid; beta-Alanine; 4-Aminobutanoic acid; Hydracrylic acid |
| osa00640 | Propanoate metabolism | 48 | 4 | 1 | 3 | 0.000612427 | 3.212945385 | 0.01265635 | 0.0923 | C00042; C00099; C00186; C01013 | Butanedioic acid; beta-Alanine; Lactic Acid; Hydracrylic acid |
| osa00760 | Nicotinate and nicotinamide metabolism | 55 | 4 | 1 | 3 | 0.001030256 | 2.987054978 | 0.015601015 | 0.0783 | C00042; C00049; C00153; C00334 | Butanedioic acid; L-Aspartic acid; Niacin; 4-Aminobutanoic acid |
| osa00630 | Glyoxylate and dicarboxylate metabolism | 62 | 4 | 2 | 2 | 0.00161735 | 2.791195935 | 0.02142989 | 0.0798 | C00037; C00042; C00158; C00209 | Glycine; Butanedioic acid; Citric acid; Oxalic acid |
| osa00460 | Cyanoamino acid metabolism | 45 | 3 | 3 | 0 | 0.005997928 | 2.221998782 | 0.06762939 | 0.0777 | C00037; C00049; C00079 | Glycine; L-Aspartic acid; DL-Phenylalanine |
| osa00052 | Galactose metabolism | 46 | 3 | 1 | 2 | 0.006380131 | 2.195170397 | 0.06762939 | 0.1091 | C00116; C00137; C00880 | Glycerol; Myo-Inositol; D-Galactonic acid |
注:pathway_id,目标通路在KEGG数据库中的ID号;pathway_name,目标通路名称;Total,目标代谢通路中代谢物的总数;Hits,目标代谢通路中总体差异代谢物数量;Up_hits:目标代谢通路中上调差异代谢物数量;Down_hits:目标代谢通路中下调差异代谢物数量;Pvalue,超几何分布检验的P值,P值越小,代表检测到的差异代谢物对该通路影响越显著;-Log10(Pvalue):P值的-log10值;FDR,假阳性校正后值,采用BH(Benjaminiand Hochberg)法计算,数值越大假阳性的可能越高;Impact,代谢通路影响值,越大说明本次检测出的差异代谢物对目标通路的影响越大。compounds:目标通路中差异代谢物的KEGG编号;Compounds_name:目标通路中差异代谢物的名称;links:KEGG通路图链接。